МЕТОДЫ КОНФОРМАЦИОННАЯ ПОДВИЖНОСТЬ
реклама

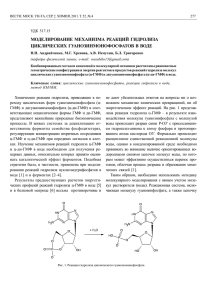
ÂÅÑÒÍ. ÌÎÑÊ. ÓÍ-ÒÀ. ÑÅÐ. 16. ÁÈÎËÎÃÈß. 2015. ¹ 3 49 ÌÅÒÎÄÛ ÓÄÊ 577.323 ÊÎÍÔÎÐÌÀÖÈÎÍÍÀß ÏÎÄÂÈÆÍÎÑÒÜ ÍÓÊËÅÎÑÎÌÛ: ÈÑÑËÅÄÎÂÀÍÈÅ ÌÅÒÎÄÎÌ ÌÎËÅÊÓËßÐÍÎÉ ÄÈÍÀÌÈÊÈ Ã.À. Àðìååâ, Ê.Â. Øàéòàí, À.Ê. Øàéòàí (êàôåäðà áèîèíæåíåðèè; e-mail: alex@molsim.org) Èññëåäîâàíèå ïðèíöèïîâ ðàáîòû õðîìàòèíà — îäíà èç êðàåóãîëüíûõ çàäà÷ ìîëåêóëÿðíîé áèîëîãèè, ñâÿçàííàÿ ñ ïîíèìàíèåì ïðîöåññîâ ýïèãåíåòè÷åñêîé ðåãóëÿöèè ýêñïðåññèè ãåíîâ, ðåïàðàöèè ÄÍÊ, ìåõàíèçìîâ íàñëåäñòâåííîñòè. Ýëåìåíòàðíîé åäèíèöåé îðãàíèçàöèè õðîìàòèíà ÿâëÿåòñÿ íóêëåîñîìà — êîìïëåêñ ÄÍÊ è áåëêîâ ãèñòîíîâ. Ñòðóêòóðà íóêëåîñîìû, ïîëó÷åííàÿ ìåòîäàìè ðåíòãåíîâñêîé êðèñòàëëîãðàôèè, íåêîòîðîå âðåìÿ ðàññìàòðèâàëàñü êàê êîíå÷íîå îñíîâíîå êîíôîðìàöèîííîå ñîñòîÿíèå íóêëåîñîìû, îäíàêî âñå áîëüøå ýêñïåðèìåíòàëüíûõ äàííûõ óêàçûâàþò íà òî, ÷òî êîíôîðìàöèîííûé ïîëèìîðôèçì è äèíàìèêà íóêëåîñîì íåïîñðåäñòâåííî âëèÿþò íà ôóíêöèîíèðîâàíèå õðîìàòèíà. Òåì íå ìåíåå îòñóòñòâóåò äåòàëüíîå ïîíèìàíèå êîíôîðìàöèîííîé äèíàìèêè íóêëåîñîì íà àòîìèñòè÷åñêîì óðîâíå.  äàííîé ðàáîòå ìû ïîäîøëè ê ðåøåíèþ ýòîé çàäà÷è ïóòåì ìîäåëèðîâàíèÿ íóêëåîñîìû ìåòîäîì ìîëåêóëÿðíîé äèíàìèêè â ÿâíîì ðàñòâîðèòåëå. Èññëåäîâàëàñü äèíàìèêà íóêëåîñîìû íà âðåìåííîì ïðîìåæóòêå 500 íñ. Ïðîèçâåäåí êîâàðèàöèîííûé àíàëèç òðàåêòîðèè ìîëåêóëÿðíîé äèíàìèêè ñèñòåìû ñ ïîñëåäóþùèì èññëåäîâàíèåì îáíàðóæåííûõ êîëëåêòèâíûõ äâèæåíèé. Èçó÷åíî ðàñïðåäåëåíèå ìîëåêóë âîäû â ñèñòåìå, ïðåäïîëàãàåòñÿ ðîëü âíóòðèíóêëåîñîìàëüíîé âîäû. Êëþ÷åâûå ñëîâà: íóêëåîñîìà, õðîìàòèí, ìîëåêóëÿðíàÿ äèíàìèêà, ýïèãåíåòèêà. Ãåíåòè÷åñêàÿ èíôîðìàöèÿ, çàêîäèðîâàííàÿ â âèäå ïîñëåäîâàòåëüíîñòè íóêëåîòèäîâ â ëèíåéíîé ìîëåêóëå ÄÍÊ, ñóììàðíàÿ äëèíà êîòîðîé â ðàçâåðíóòîì ñîñòîÿíèè ìîæåò äîñòèãàòü íåñêîëüêèõ ìåòðîâ, ðàçìåùàåòñÿ â ýóêàðèîòè÷åñêèõ êëåòêàõ â âåñüìà îãðàíè÷åííîé îáëàñòè ïðîñòðàíñòâà — ÿäðå êëåòêè, ðàçìåðû êîòîðîãî íå ïðåâûøàþò 10 ìêì. Êîìïàêòèçàöèÿ ìîëåêóë ÄÍÊ ïðè îäíîâðåìåííîì îáåñïå÷åíèè äîñòóïà áåëêîâûõ ôàêòîðîâ ê íåîáõîäèìûì ó÷àñòêàì ãåíîìà è êîìïëåêñíàÿ ðåãóëÿöèÿ äîñòóïà — îñíîâíûå ôóíêöèè õðîìàòèíà. Íà ïåðâîì ýòàïå êîìïàêòèçàöèè ÄÍÊ íàèáîëüøåå çíà÷åíèå èãðàåò âçàèìîäåéñòâèå ÄÍÊ ñ áåëêàìè-ãèñòîíàìè è ôîðìèðîâàíèå íóêëåîñîì. Ïîíèìàíèå ñòðóêòóðû è îðãàíèçàöèè âçàèìîäåéñòâèé â íóêëåîñîìàõ ÿâëÿåòñÿ âàæíûì ýòàïîì íà ïóòè èçó÷åíèÿ ñòðóêòóðû è äèíàìèêè õðîìàòèíà. Ïðè îïèñàíèè ñòðóêòóðû õðîìàòèíà ïðèíÿòî èñïîëüçîâàòü ñëåäóþùèå òåðìèíû: ÿäðî íóêëåîñîìíîé ÷àñòèöû (nucleosome core particle) — êîìïëåêñ èç âîñüìè ãèñòîíîâ è 145—147 ï.í. Òåðìèí íóêëåîñîìà ïðèíÿòî ïîíèìàòü êàê ÿäðî íóêëåîñîìíîé ÷àñòèöû âìåñòå ñ ñîåäèíÿþùèìè ñîñåäíèå íóêëåîñîìû ó÷àñòêàìè ÄÍÊ. Íóêëåîñîìó, ñîäåðæàùóþ ãèñòîí H1, íàçûâàþò õðîìàòîñîìîé. Äàëåå ïî òåêñòó òåðìèíû íóêëåîñîìà è ÿäðî íóêëåîñîìíîé ÷àñòèöû ñëåäóåò ñ÷èòàòü ñèíîíèìè÷íûìè. Ñòðóêòóðà íóêëåîñîì äîëãîå âðåìÿ ïîñëå èõ îòêðûòèÿ â 1974 ã. Ð. Êîðíáåðãîì [1] îñòàâàëàñü íåÿñ- íîé, áûëî ïîñòðîåíî ìíîæåñòâî ìîäåëåé, îò áëèçêèõ ê ñîâðåìåííûì ïðåäñòàâëåíèÿì äî äîâîëüíî ýêñòðàâàãàíòíûõ. Äåáàòû ïî ïîâîäó ñòðóêòóðû ïðîäîëæàëèñü âïëîòü äî 1997 ã., êîãäà ìåòîäàìè ðåíòãåíîâñêîé êðèñòàëëîãðàôèè áûëà îïðåäåëåíà ïåðâàÿ ñòðóêòóðà íóêëåîñîìû ñ ïî÷òè àòîìàðíûì ðàçðåøåíèåì [2]. Íóêëåîñîìà ïðåäñòàâëÿåò ñîáîé îêòàìåð áåëêîâ ãèñòîíîâ, êîòîðûé íåñåò íà ñåáå 145—147 íóêëåîòèäíûõ ïàð. ÄÍÊ çàêðó÷åíà âîêðóã îêòàìåðà, îáðàçóÿ 1,65 âèòêà ëåâîçàêðó÷åííîé ñóïåðñïèðàëè, ò.å. ÄÍÊ îòðèöàòåëüíî ñâåðõñïèðàëèçîâàíà. Áåëêîâîå ÿäðî íóêëåîñîìû îáðàçóåò öèëèíäð äèàìåòðîì 65 C è âûñîòîé 60 C. Äëÿ íóêëåîñîìû õàðàêòåðíà îñü ïñåâäîñèììåòðèè — äèàäíàÿ îñü.  ÿäðî íóêëåîñîìû âõîäèò ÷åòûðå ñåìåéñòâà ãèñòîíîâ H3, H4, H2A è H2B, êàæäûé èç ãèñòîíîâ ïðåäñòàâëåí äâàæäû. Âîñåìü ãèñòîíîâ íóêëåîñîìû ñîáðàíû â ÷åòûðå ãåòåðîäèìåðà: äâà H3-H4 è äâà H2A-H2B. Êàæäîå ñåìåéñòâî ãèñòîíîâ âêëþ÷àåò â ñåáÿ ìíîæåñòâî ãèñòîíîâûõ âàðèàíòîâ, íåñìîòðÿ íà òî ÷òî ãèñòîíû ÿâëÿþòñÿ îäíèìè èç ñàìûõ êîíñåðâàòèâíûõ áåëêîâ íàðàâíå ñ ðèáîñîìàëüíûìè áåëêàìè. Íàïðèìåð, â ÿäðàõ êëåòîê Arabidopsis arenosa îáíàðóæåíî 13 âàðèàíòîâ H2A, 11 — H2B, 9 — H3 è 1 — H4, çà÷àñòóþ îäíó ôîðìó ãèñòîíà êîäèðóþò íåñêîëüêî ãåíîâ [3], íàõîäÿùèõñÿ â òàê íàçûâàåìûõ ãèñòîíîâûõ êëàñòåðàõ. Íà 145—147 ï.í., ðàçìåùåííûõ íà ãèñòîííîì îêòàìåðå, ïðèõîäèòñÿ 14 ñàéòîâ ñâÿçûâàíèÿ. Êîí- 50 òàêòû ÄÍÊ ñ ãèñòîíàìè îáåñïå÷èâàþòñÿ â îñíîâíîì çà ñ÷åò ôîñôîäèýôèðíûõ ãðóïïèðîâîê ñàõàðîôîñôàòíîãî îñòîâà, òàêèì îáðàçîì, îáðàçîâàíèå êîíòàêòîâ ïðÿìî íå çàâèñèò îò ïîñëåäîâàòåëüíîñòè. Ñáîðêà íóêëåîñîì â æèâîé êëåòêå ïðîèñõîäèò ïðè ïîìîùè ñëîæíîé ñèñòåìû áåëêîâ øàïåðîíîâ [4], êîòîðûå òàêæå ìîãóò áûòü ñïåöèôè÷íû äëÿ ðàçëè÷íûõ âàðèàíòîâ ãèñòîíîâ. Íóêëåîñîìà — ïîäâèæíàÿ ñòðóêòóðà, êîòîðîé ñâîéñòâåííû êàê âûñîêî÷àñòîòíûå òåïëîâûå äâèæåíèÿ, òàê è êðóïíîìàñøòàáíûå êîíôîðìàöèîííûå ïåðåñòðîéêè. Èñõîäÿ èç ðàñïðåäåëåíèÿ B-ôàêòîðîâ â êðèñòàëëè÷åñêèõ ñòðóêòóðàõ, ñëåäóåò îòìåòèòü, ÷òî ïîäâèæíîñòü ÄÍÊ çíà÷èòåëüíî âûøå, íåæåëè ïîäâèæíîñòü áåëêîâîé ñîñòàâëÿþùåé íóêëåîñîìû.  òî æå âðåìÿ íàõîäÿùèåñÿ â ìàëûõ áîðîçäêàõ ÄÍÊ àðãèíèíû ïðåïÿòñòâóþò ñêîëüæåíèþ ñïèðàëè ÄÍÊ íà ãèñòîíàõ, îãðàíè÷èâàÿ åå ìîáèëüíîñòü [5]. Ìàñøòàáíóþ ïîäâèæíîñòü íóêëåîñîì ïðèíÿòî ðàçäåëÿòü íà “äûõàíèå” (breathing), ðàçìàòûâàíèå ÄÍÊ (unwinding) è ðàñêðûâàíèå (gapping) [6]. Ïåðâûå äâà òèïà êîíôîðìàöèîííîé ïîäâèæíîñòè îïèñûâàþò ÷àñòè÷íóþ äèññîöèàöèþ ÄÍÊ îò ãèñòîíîâ, ïîä “äûõàíèåì” ïîíèìàþò ñèììåòðè÷íîå ðàçìàòûâàíèå ÄÍÊ. Òàêèå äâèæåíèÿ ïîòåíöèàëüíî îáëåã÷àþò âõîæäåíèå ïîëèìåðàç â íóêëåîñîìó è ïîêàçàíû ýêñïåðèìåíòàëüíî [7]. Èçâåñòíî, ÷òî ðàñêðó÷èâàíèå è “äûõàíèå” íóêëåîñîìû â ïðèñóòñòâèè ôàêòîðà ïåðåñòðîéêè õðîìàòèíà FACT (FAcilitates Chromatin Transcription) ïðèâîäèò ê ñòàáèëèçàöèè “ðàñêðûòûõ” ñîñòîÿíèé, ïîâûøàÿ äîñòóïíîñòü íóêëåîñîì äëÿ ïîëèìåðàç [8]. Òðåòüå äâèæåíèå — ðàñêðûâàíèå — áûëî ïðåäñêàçàíî òåîðåòè÷åñêè. Ïîëàãàåòñÿ, ÷òî òàêîé òèï äâèæåíèé ìîæåò èãðàòü ðîëü â îïðåäåëåíèè ïëîòíîñòè óïàêîâêè 30 íì ôèáðèëëû ïðè êîëëåêòèâíîì äâèæåíèè âñåõ íóêëåîñîì [9]. Ïîíèìàíèå ïðèíöèïîâ ðàáîòû õðîìàòèíà — ñëåäóþùàÿ áîëüøàÿ çàäà÷à ìîëåêóëÿðíîé áèîëîãèè, ñðàâíèìàÿ ñ çàäà÷åé ðàñøèôðîâêè ãåíîìà. Íà óðîâíå íóêëåîñîì îñóùåñòâëÿåòñÿ áîëüøîå êîëè÷åñòâî ðàçëè÷íûõ ìåõàíèçìîâ ðåãóëÿöèè ýêñïðåññèè ãåíîâ. Îäèí èç òàêèõ ìåõàíèçìîâ — òàê íàçûâàåìûé íóêëåîñîìàëüíûé áàðüåð [10]. Ïðè ïðèáëèæåíèè ÐÍÊ-ïîëèìåðàçû íóêëåîñîìà äîëæíà ïðåòåðïåâàòü êîíôîðìàöèîííûå èçìåíåíèÿ, äëÿ òîãî ÷òîáû ñäåëàòü ÄÍÊ äîñòóïíîé.  ÷àñòíîñòè, èçâåñòíû ìóòàöèè (SIN ìóòàíòû), êîòîðûå îáëåã÷àþò òðàíñêðèïöèþ [11]. Ðàçëè÷íûå ïîñëåäîâàòåëüíîñòè èìåþò ðàçíîå ñðîäñòâî ê ãèñòîíàì è ìîäóëèðóþò âåðîÿòíîñòü ïðîõîæäåíèÿ ïîëèìåðàçû. Èçó÷åíèå ìåæìîëåêóëÿðíûõ âçàèìîäåéñòâèé íà àòîìèñòè÷åñêîì óðîâíå è ïîíèìàíèå ýíåðãåòèêè äàííûõ âçàèìîäåéñòâèé, èõ ôóíêöèîíàëüíîé çíà÷èìîñòè ÿâëÿåòñÿ îäíîé èç îñíîâíûõ çàäà÷ ìîëåêóëÿðíîé áèîëîãèè.  ïîñëåäíåå âðåìÿ â êîìïëåêñå ìåòîäîâ èññëåäîâàíèé íàðàâíå ñ òðàäèöè- ÂÅÑÒÍ. ÌÎÑÊ. ÓÍ-ÒÀ. ÑÅÐ. 16. ÁÈÎËÎÃÈß. 2015. ¹ 3 îííûìè ýêñïåðèìåíòàëüíûìè ìåòîäàìè (âêëþ÷àÿ ðåíòãåíîâñêóþ êðèñòàëëîãðàôèþ, FRET, ChIP-Seq, ðàçëè÷íûå ìåòîäû àíàëèçà ñêîðîñòè òðàíñêðèïöèè è äð.) âàæíóþ ðîëü ñòàëè èãðàòü ìåòîäû ñòðóêòóðíîãî è äèíàìè÷åñêîãî êîìïüþòåðíîãî ìîäåëèðîâàíèÿ.  ÷àñòíîñòè, ìåòîä ìîëåêóëÿðíîé äèíàìèêè ðàññìàòðèâàåòñÿ êàê ñâîåîáðàçíûé “âû÷èñëèòåëüíûé ìèêðîñêîï” [12]. Íà îñíîâå “çàìîðîæåííûõ” ñòðóêòóð áèîìàêðîìîëåêóë, ïîëó÷àåìûõ ìåòîäàìè ðåíòãåíîâñêîé êðèñòàëëîãðàôèè, ìîëåêóëÿðíàÿ äèíàìèêà (ÌÄ) ïîçâîëÿåò ïîñòðîèòü äèíàìè÷åñêóþ ìîäåëü, èññëåäîâàòü åå äâèæåíèÿ è êîíôîðìàöèîííûå èçìåíåíèÿ. Ìåòîäû êðèñòàëëîãðàôèè íå ëèøåíû íåòî÷íîñòåé, ñâÿçàííûõ ñ ïëîòíîé óïàêîâêîé è îòáîðîì êîíôîðìåðîâ, ñîâìåñòèìûõ ñ ýòîé óïàêîâêîé. Òàêæå ñëåäóåò îòìåòèòü, ÷òî ÌÄ ïîçâîëÿåò ðåøàòü òàêèå çàäà÷è, êàê èññëåäîâàíèå ðàñïðåäåëåíèÿ èîíîâ è âîäû âîêðóã áèîìîëåêóë, êîòîðûå ñ òðóäîì ðåøàþòñÿ ìåòîäàìè êðèñòàëëîãðàôèè.  ýòîé ñâÿçè îñíîâíîé öåëüþ äàííîé ðàáîòû ÿâëÿëîñü èññëåäîâàíèå ñòðóêòóðíûõ è äèíàìè÷åñêèõ õàðàêòåðèñòèê íóêëåîñîì ìåòîäàìè êîìïüþòåðíîãî ìîäåëèðîâàíèÿ, à òàêæå îöåíêà ðîëè ðàñòâîðèòåëÿ â ôîðìèðîâàíèè ñòðóêòóðû íóêëåîñîì. Ìàòåðèàëû è ìåòîäû Äëÿ ïðîâåäåíèÿ èññëåäîâàíèé èñïîëüçîâàëîñü ñèëîâîå ïîëå AMBER99SB-BSC0 [13, 14]. Ìîäåëü áûëà ñîçäàíà íà îñíîâàíèè êðèñòàëëè÷åñêîé ñòðóêòóðû, âçÿòîé èç áàíêà PDB ñ èíäåêñîì 1KX5 [15]. Îò íóêëåîñîì óäàëÿëèñü ïîäâèæíûå ãèñòîíîâûå “õâîñòû” ñîãëàñíî ïîçèöèÿì, âçÿòûì â ðàáîòå [16], èîíû ìàðãàíöà çàìåíÿëèñü èîíàìè ìàãíèÿ. Èñïîëüçîâàëèñü ïðÿìîóãîëüíûå ðàñ÷åòíûå ÿ÷åéêè, ðàçìåð êîòîðûõ ïîäáèðàëñÿ òàê, ÷òîáû íàèìåíüøåå ðàññòîÿíèå îò íóêëåîñîìû äî îäíîé èç ãðàíåé ÿ÷åéêè áûëî íå ìåíåå 1 íì. Âñå ñâîáîäíîå ïðîñòðàíñòâî çàïîëíÿëîñü ìîëåêóëàìè âîäû, äëÿ ìîäåëèðîâàíèÿ êîòîðîé ïðèìåíÿëàñü ìîäåëü TIP3P. Èñïîëüçîâàëèñü èîíû íàòðèÿ è õëîðà â êîíöåíòðàöèè 150 ìÌ, êîíöåíòðàöèÿ èîíîâ ðàññ÷èòûâàëàñü êàê êîëè÷åñòâî èîíîâ ê îáúåìó ðàñòâîðèòåëÿ, à íè ê îáúåìó ðàñ÷åòíîé ÿ÷åéêè. Òàêîé ñïîñîá, ïðèìåíÿëñÿ òàê êàê â ñèëó ìàëûõ ðàçìåðîâ ìîäåëèðóåìîé ñèñòåìû, ñàìà ìîëåêóëà çàíèìàåò çíà÷èòåëüíûé îáúåì ÿ÷åéêè. Îáúåì ýëåìåíòàðíîé ÿ÷åéêè ðàâåí 2100 íì3. Ðàñ÷åò òðàåêòîðèé äèíàìèêè ïðîèçâîäèëñÿ ñ øàãîì èíòåãðèðîâàíèÿ 2 ôñ. Äëÿ òîãî ÷òîáû èçáåæàòü ÷ðåçìåðíîãî ïåðåìåùåíèÿ íóêëåîñîìû â ïðîñòðàíñòâå, íà Ña àòîìû àìèíîêèñëîò ãèñòîíà H3 íàêëàäûâàëñÿ äîïîëíèòåëüíûé ãàðìîíè÷åñêèé ïîòåíöèàë, âîçâðàùàþùèé èõ íà÷àëüíîå ïîëîæåíèå. Äëÿ òîãî ÷òîáû èçáåæàòü êðàåâûõ ýôôåêòîâ, èñïîëüçîâàëèñü ïåðèîäè÷åñêèå ãðàíè÷íûå óñëîâèÿ. Ýëåêòðîñòàòè÷åñêèå âçàèìîäåéñòâèÿ ó÷èòûâàëèñü ïðè ïîìîùè ìåòîäà PME [17] ñ øàãîì ÂÅÑÒÍ. ÌÎÑÊ. ÓÍ-ÒÀ. ÑÅÐ. 16. ÁÈÎËÎÃÈß. 2015. ¹ 3 ðåøåòêè 1 C, ðàäèóñîì îáðåçàíèÿ âçàèìîäåéñòâèé â ïðÿìîì ïðîñòðàíñòâå 1 íì è êóáè÷åñêîé èíòåðïîëÿöèåé. Äëÿ ó÷åòà âàí-äåð-âààëüñîâûõ âçàèìîäåéñòâèé èñïîëüçîâàëñÿ ðàäèóñ îáðåçàíèÿ â 1 íì ñ ïëàâíûì âûðàâíèâàíèåì ïîòåíöèàëà íà ãðàíèöå îáðåçàíèÿ. Äëÿ òåðìîñòàòèðîâàíèÿ ðàñ÷åòíîé ÿ÷åéêè ïðèìåíÿëñÿ ìîäèôèöèðîâàííûé òåðìîñòàò Áåðåíäñåíà ñî ñòîõàñòè÷åñêîé ïðèáàâêîé [18], âîäà è ìàêðîìîëåêóëà òåðìîñòàòèðîâàëèñü ðàçäåëüíî.  êà÷åñòâå áàðîñòàòà ïðèìåíÿëñÿ èçîòðîïíûé áàðîñòàò Ïàðèíåëëî—Ðàìàíà [19]. Äëÿ èññëåäîâàíèÿ êðóïíîìàñøòàáíûõ ñêîððåëèðîâàííûõ äâèæåíèé íóêëåîñîìû èñïîëüçîâàëñÿ ìåòîä êîâàðèàöèîííîãî àíàëèçà, à òàêæå ìåòîä ðàñ÷åòà íàèìåíüøåãî ñðåäíåêâàäðàòè÷íîãî îòêëîíåíèÿ ñòðóêòóðû ïî òðàåêòîðèè (RMSD). Ðàñ÷åòû ïðîèçâîäèëèñü â ïðîãðàììå Gromacs 4.5.4 [20] íà ñóïåðêîìïüþòåðå ÌÃÓ “Ëîìîíîñîâ” [21] ñ èñïîëüçîâàíèåì îò 64 äî 1024 ïðîöåññîðîâ. Îáðàáîòêà òðàåêòîðèé ïðîèçâîäèëàñü ïðè ïîìîùè âñòðîåííûõ â ïàêåò Gromacs óòèëèò è ñîáñòâåííûõ èñïîëíÿåìûõ ôàéëîâ, íàïèñàííûõ íà ÿçûêàõ Python è TCL íà âû÷èñëèòåëüíûõ êëàñòåðàõ êàôåäðû áèîèíæåíåðèè. Äëÿ âèçóàëèçàöèè òðàåêòîðèé è ìîäåëåé ïðèìåíÿëàñü ïðîãðàììà VMD 1.9.1. [22]. Ðåçóëüòàòû è îáñóæäåíèå Ìîäåëèðîâàíèå íà ïðîòÿæåíèè 500 íñ ïîêàçàëî, ÷òî ñèñòåìà â öåëîì ìàëî èçìåíÿåò ñâîþ ôîðìó, ïðè ýòîì ïîäâèæíîñòü ÄÍÊ çíà÷èòåëüíî âûøå òàêîâîé ó ãèñòîíîâ, ÷òî ñîâïàäàåò ñ ýêñïåðèìåí- 51 òàëüíûìè äàííûìè. Íàáîð êîíôîðìàöèé, êîòîðûå íóêëåîñîìà ïðèíèìàåò â òå÷åíèè ÌÄ, ïîêàçàí íà ðèñóíêå. Ðàñïðåäåëåíèå ðàññ÷èòàííûõ B-ôàêòîðîâ âäîëü ïîñëåäîâàòåëüíîñòè ÄÍÊ îêàçàëîñü ïîõîæå íà ðàñïðåäåëåíèå â êðèñòàëëè÷åñêîé ñòðóêòóðå, íî ïîäâèæíîñòü ÄÍÊ â ðàñ÷åòíîé ñèñòåìå çíà÷èòåëüíî âûøå, òàê êàê ìîäåëèðîâàíèå ïðîèñõîäèò ïðè òåìïåðàòóðå 300° K, à êðèñòàëë íàõîäèòñÿ â êðèîãåííîì äèàïàçîíå òåìïåðàòóð. Ðàñ÷åò B-ôàêòîðîâ äëÿ àòîìîâ â ñèñòåìå ïðîèçâîäèëñÿ ïî ôîðìóëå B = 8π2<u2> ãäå <u2> — ñðåäíåêâàäðàòè÷íîå îòêëîíåíèå êîîðäèíàò àòîìà. Äîïîëíèòåëüíî ïîêàçàíà íåðàâíîìåðíîñòü ðàñïðåäåëåíèÿ B-ôàêòîðîâ âäîëü ÄÍÊ, ÷òî ñâÿçàíî ñ áîëüøåé ìîáèëüíîñòüþ è ðàñïëåòàíèåì ÄÍÊ âáëèçè ìåñò âõîäà â íóêëåîñîìó. Èç-çà âûñîêîé ïîäâèæíîñòè ÄÍÊ êîëè÷åñòâî êîíòàêòîâ, îáðàçóåìûõ ÄÍÊ ñ ãèñòîíàìè, ïîñòîÿííî èçìåíÿåòñÿ è â ñðåäíåì îòëè÷àåòñÿ îò ÷èñëà êîíòàêòîâ â êðèñòàëëè÷åñêîé ñòðóêòóðå. Íàèáîëåå çàìåòíûå îòëè÷èÿ íàáëþäàþòñÿ â ïîçèöèè +15—+35 îò âõîäà â íóêëåîñîìó, êîíôîðìàöèÿ ÄÍÊ â êðèñòàëëè÷åñêîé ñòðóêòóðå, ïî âñåé âèäèìîñòè, íàïðÿæåíà è â õîäå äèíàìèêè ïðèõîäèò â ðàâíîâåñíîå ñîñòîÿíèå. Òàêæå ñòîèò îòìåòèòü, ÷òî â êðèñòàëëè÷åñêîé ñòðóêòóðå ìåæäó êîíòàêòàìè èìåþòñÿ ó÷àñòêè ÄÍÊ, íå ñâÿçàííûå ñ ãèñòîíàìè. Êîëè÷åñòâî òàêèõ ó÷àñòêîâ â óñðåäíåííîé ïî òðàåêòîðèè ñòðóêòóðå ìåíüøå, à ñàìè ó÷àñòêè îòëè÷àþòñÿ ìåíüøåé ïðîòÿæåííîñòüþ. Òàêèì îáðàçîì, èñïîëüçîâàíèå Ãèñòîíû îòîáðàæåíû â âèäå âòîðè÷íîé ñòðóêòóðû, ÄÍÊ ïîêàçàíà â âèäå ëèíèé 52 êðèñòàëëè÷åñêîé ñòðóêòóðû äëÿ ïîñòðîåíèÿ ìîäåëè ñâÿçûâàíèÿ ÄÍÊ ñ ãèñòîíàìè íå â ïîëíîé ìåðå âîñïðîèçâîäèò âêëàä êàæäîé ïàðû íóêëåîòèäîâ â ôîðìèðîâàíèå íóêëåîñîìû. Êîâàðèàöèîííûé àíàëèç äâèæåíèé àòîìîâ â ÌÄ, ïðîâåäåííûé ïî áåëêîâîìó îñòîâó ãèñòîíîâ è îñòîâó ÄÍÊ, âûÿâèë íàáîð êðóïíîìàñøòàáíûõ äâèæåíèé â íóêëåîñîìå. Íàèáîëåå àìïëèòóäíûé ñîáñòâåííûé âåêòîð, ïîëó÷åííûé â ðåçóëüòàòå êîâàðèàöèîííîãî àíàëèçà, ìîæíî êëàññèôèöèðîâàòü êàê äâèæåíèå ðàñêðûâàíèÿ íóêëåîñîìû.  ðàáîòå [9] ñîñòîÿíèå îòêðûòîé íóêëåîñîìû ïîëó÷èëè èñêóññòâåííî, ïóòåì ôèêñàöèè êîíòàêòà ãèñòîíîâ H3-H3 â êà÷åñòâå “ïåòëè” è ââåäåíèÿ äîïîëíèòåëüíîãî ïîòåíöèàëà äëÿ ðàñêðûòèÿ “ïåòëè”. Êîëè÷åñòâî ýíåðãèè, íåîáõîäèìîé äëÿ òîãî ÷òîáû ðàñêðûòü íóêëåîñîìó (30 êêàë/ìîëü), ñëèøêîì âåëèêî äëÿ ñïîíòàííîãî ðàñêðûâàíèÿ íóêëåîñîìû. Òåì íå ìåíåå ñ÷èòàåòñÿ, ÷òî òàêîå äâèæåíèå âîçìîæíî ïðè óñëîâèè êîëëåêòèâíîãî ïåðåõîäà öåëîé öåïè íóêëåîñîì [9]. Îáíàðóæåíèå äàííîé ìîäû äâèæåíèé â òðàåêòîðèè ñâîáîäíîé äèíàìèêè ñâèäåòåëüñòâóåò î òîì, ÷òî äâèæåíèÿ, âåäóùèå ê ðàñêðûòèþ íóêëåîñîìû, âîçìîæíû, íî, ñóäÿ ïî èõ ìàëîé àìïëèòóäå, ìàëîâåðîÿòíû áåç íàëè÷èÿ âíåøíèõ ñèë, ñïîñîáñòâóþùèõ êîíôîðìàöèîííîìó ïåðåõîäó. Âîçìîæíîå ôóíêöèîíàëüíîå çíà÷åíèå äâèæåíèÿ ðàñêðûâàíèÿ íóêëåîñîì íåèçâåñòíî îäíîçíà÷íî, ïðåäïîëîæèòåëüíî, îíî ìîæåò èãðàòü ðîëü â ïðîöåññå êîìïàêòèçàöèè 30-íàíîìåòðîâîé ôèáðèëëû [9]. Âåñüìà âåðîÿòíîé âèäèòñÿ âîçìîæíîñòü îòêðûâàíèÿ íóêëåîñîì ïðè âçàèìîäåéñòâèè ÐÍÊïîëèìåðàçû II ñî âòîðîé ïåòëåé ÄÍÊ. Ýíåðãèÿ, êîòîðóþ íåîáõîäèìî çàòðàòèòü äëÿ òîãî, ÷òîáû ðàñêðûòü íóêëåîñîìó, ñîñòîèò èç ýíåðãèè ãèäðàòàöèè íåïîëÿðíûõ ïðèëåãàþùèõ ó÷àñòêîâ ãèñòîíîâ H2A-H2B è ýíåðãèè äåôîðìàöèè ñóïåðñïèðàëè ÄÍÊ. Òàêèì îáðàçîì, íóêëåîñîìû ñ ãèñòîííûìè âàðèàíòàìè, ñîäåðæàùèìè äðóãèå àìèíîêèñëîòíûå îñòàòêè â ïîëîæåíèè êîíòàêòîâ, ìîãóò îòëè÷àòüñÿ âåðîÿòíîñòüþ îòêðûâàíèÿ, ÷òî ìîæåò ïðèâåñòè ê óêîðà÷èâàíèþ èëè óäëèíåíèþ ïàóçû ïðè ïðîõîæäåíèè ÐÍÊ-ïîëèìåðàçû. Âûÿâëåííîå äâèæåíèå ïðîèñõîäèò ñ äîâîëüíî íèçêîé äëÿ âðåìåí ìîäåëèðîâàíèÿ ÷àñòîòîé, çà 500 íñ ñèñòåìà ïðåòåðïåâàåò òàêèå äâèæåíèÿ îäíîêðàòíî, ÷òî ñâèäåòåëüñòâóåò î òîì, ÷òî ðàñ÷åò íå äîñòàòî÷åí äëÿ îáíàðóæåíèÿ âñåõ ìîä äâèæåíèé. Çíà÷èòåëüíîãî óâåëè÷åíèÿ âðåìåíè ìîäåëèðîâàíèÿ íå ïîëó÷èòñÿ äîáèòüñÿ ïóòåì óâåëè÷åíèÿ ÷èñëà âû÷èñëèòåëüíûõ óçëîâ òåêóùåé àðõèòåêòóðû, òàê êàê ðîñò ïðîèçâîäèòåëüíîñòè ñèñòåìû ñèëüíî îãðàíè÷åí øèíîé îáìåíà äàííûìè ìåæäó óçëàìè. Äëÿ óâåëè÷åíèÿ âðåìåíè ìîäåëèðîâàíèÿ ìîæíî èñïîëüçîâàòü âû÷èñëèòåëüíûå ìàøèíû, ñîçäàííûå ñïåöèàëüíî äëÿ ðåøåíèÿ çàäà÷ ìîëåêóëÿðíîé äèíàìèêè, íàïðèìåð êîìïüþòåð “Anton” [23], ÂÅÑÒÍ. ÌÎÑÊ. ÓÍ-ÒÀ. ÑÅÐ. 16. ÁÈÎËÎÃÈß. 2015. ¹ 3 èëè ïðèáåãàòü ê èñïîëüçîâàíèþ îãðóáëåííûõ ìîäåëåé ðàñòâîðèòåëÿ èëè ñàìèõ íóêëåîñîì. Íåñìîòðÿ íà ñòàáèëüíîñòü íóêëåîñîì è ïëîòíóþ óïàêîâêó ãèñòîíîâ â äèìåðû, îêòàìåð ãèñòîíîâ óïàêîâàí íåïëîòíî è ñîäåðæèò â ñåáå áîëüøîå êîëè÷åñòâî ìîëåêóë âîäû, êîòîðûå ôîðìèðóþò ñâîåîáðàçíûå âîäÿíûå “ìîñòèêè” â íóêëåîñîìå. Êîëè÷åñòâî âîäû â ñîñòàâå íóêëåîñîìû äîâîëüíî çíà÷èòåëüíî èçìåíÿåòñÿ íà ïðîòÿæåíèè âðåìåíè ìîäåëèðîâàíèÿ è ïðåâîñõîäèò êîëè÷åñòâî âîäû â êðèñòàëëè÷åñêîé ñòðóêòóðå. Íàèáîëüøèå èçìåíåíèÿ êîëè÷åñòâà õàðàêòåðíû äëÿ íàõîäÿùåéñÿ â íóêëåîñîìíîé ïîðå âîäû, â òî âðåìÿ êàê êîëè÷åñòâî ìîëåêóë, íàõîäÿùèõñÿ âáëèçè êîíòàêòîâ äèìåðîâ ãèñòîíîâ, ñîõðàíÿåòñÿ íåèçìåííûì. Íàèìåíüøåå êîëè÷åñòâî ìîëåêóë âîäû (â ñðåäíåì 63 ìîëåêóëû) õàðàêòåðíî äëÿ êîíòàêòà äèìåðîâ ãèñòîíîâ H3-H4, ôîðìèðóþùèõ òåòðàìåð — íóêëåîñîìíîå ÿäðî, â òî âðåìÿ êàê îñòàëüíûå êîíòàêòû äèìåðîâ ñîäåðæàò ïðèáëèçèòåëüíî îäèíàêîâîå êîëè÷åñòâî ìîëåêóë âîäû (îêîëî 100 ìîëåêóë). Áîëüøåå ÷èñëî ìîëåêóë âîäû íà ó÷àñòêå âçàèìîäåéñòâèÿ äèìåðîâ ãèñòîíîâ ñâèäåòåëüñòâóåò î ìåíåå ïëîòíîì êîíòàêòå, ÷òî íå ïðîòèâîðå÷èò ýêñïåðèìåíòàëüíûì äàííûì, òàê êàê ãèñòîíû H2A-H2B óäàëÿþòñÿ èç íóêëåîñîìû ëåã÷å âñåãî. Íàëè÷èå â íóêëåîñîìå áîëüøîãî ÷èñëà ìîëåêóë âîäû ìåæäó ãèñòîíàìè äåëàåò åå ïðîíèöàåìîé äëÿ îäíîâàëåíòíûõ èîíîâ, ïîçâîëÿÿ èì ýôôåêòèâíî ýêðàíèðîâàòü çàðÿæåííûå ãðóïïèðîâêè ãèñòîíîâ è ÄÍÊ. Íàèáîëåå âåðîÿòíûì íàçíà÷åíèåì âîäû â íóêëåîñîìíîé ïîðå ÿâëÿåòñÿ ñíèæåíèå ñèëû çàðÿäîâûõ âçàèìîäåéñòâèé ìåæäó ãèñòîíàìè, íåñóùèìè çíà÷èòåëüíûé ïîëîæèòåëüíûé çàðÿä çà ñ÷åò ïîâûøåííîé äèýëåêòðè÷åñêîé ïðîíèöàåìîñòè. Ïîìèìî ýòîãî âîäà â íóêëåîñîìíîé ïîðå ïîçâîëÿåò èîíàì ñâîáîäíî ïðîíèêàòü ñêâîçü íóêëåîñîìó è ýêðàíèðîâàòü çàðÿäû íà ïîâåðõíîñòè ãèñòîíîâ.  çàêëþ÷åíèè ðàáîòû ìîæíî ñäåëàòü ñëåäóþùèå âûâîäû: ñòðóêòóðà íóêëåîñîìû â öåëîì ñòàáèëüíà íà âðåìåíàõ ïîðÿäêà 500 íñ, à ñðåäè êðóïíîìàñøòàáíûõ êîëëåêòèâíûõ äâèæåíèé âûäåëÿåòñÿ ìîäà äâèæåíèé, ïðè êîòîðîé äâà ñóïåðâèòêà ÄÍÊ (â ìåñòå ïåðåñå÷åíèÿ ñ äèàäíîé îñüþ íóêëåîñîìû) è ñâÿçàííûå ñ íèìè îáëàñòè ãèñòîíîâ ïðèáëèæàþòñÿ è óäàëÿþòñÿ äðóã îò äðóãà. Äàííàÿ ìîäà äâèæåíèé ïîòåíöèàëüíî ñâÿçàíà ñ ïðîöåññàìè ïðîõîæäåíèÿ òðàíñêðèïöèè è ïëîòíîé êîìïàêòèçàöèè õðîìàòèíà. Íóêëåîñîìà ñîäåðæèò â ñâîåé ñòðóêòóðå áîëüøîå êîëè÷åñòâî ìîëåêóë âîäû, â íóêëåîñîìàëüíîé ïîðå â ñðåäíåì ñîäåðæèòñÿ 178 ìîëåêóë âîäû, êîëè÷åñòâî ìîëåêóë âîäû â êîíòàêòàõ äèìåðîâ ãèñòîíîâ âàðüèðóåò îò 62 äî 105.  îáðàçîâàíèè êîíòàêòà ìåæäó äâóìÿ äèìåðàìè ãèñòîíîâ H3-H4 ó÷àñòâóåò ìåíüøåå êîëè÷åñòâî ìîëåêóë âîäû (62), ÷åì ïðè îáðàçîâàíèè àíàëîãè÷íîãî êîíòàêòà ìåæäó äèìåðàìè H3-H4 è H2À-H2B (102—105). ÂÅÑÒÍ. ÌÎÑÊ. ÓÍ-ÒÀ. ÑÅÐ. 16. ÁÈÎËÎÃÈß. 2015. ¹ 3 Âñå âû÷èñëåíèÿ ïðîèçâåäåíû ñ èñïîëüçîâàíèåì ñóïåðêîìïüþòåðíîãî êîìïëåêñà ÌÃÓ èìåíè Ì.Â. Ëîìîíîñîâà [21]. 53 *** Äàííàÿ ðàáîòà âûïîëíåíà ïðè ïîääåðæêå ãðàíòà Ðîññèéñêîãî íàó÷íîãî ôîíäà (¹ 14-24-00031). ÑÏÈÑÎÊ ËÈÒÅÐÀÒÓÐÛ 1. Kornberg R.D. Chromatin structure: A repeating unit of histones and DNA // Science. 1974. Vol. 184. N 4139. P. 868—871. 2. Luger K., Mader A.W., Richmond R.K., Sargent D.F., Richmond T.J. Crystal structure of the nucleosome core particle at 2.8 C a resolution // Nature. 1997. Vol. 389. N 6648. P. 251—260. 3. Talbert P.B., Ahmad K., Almouzni G. A unified phylogeny-based nomenclature for histone variants // Epigenet. Chromatin. 2012. Vol. 5. N 1. P. 7. 4. Burgess R.J., Zhang Z. Histone chaperones in nucleosome assembly and human disease // Nat. Struct. Mol. Biol. 2013. Vol. 20. N 1. P. 14—22. 5. Biswas M., Langowski J., Bishop T.C. Atomistic simulations of nucleosomes // Wiley Interdiscip Rev Comput Mol Sci. 2013. Vol. 3. N 4. P. 378—392. 6. Zlatanova J., Bishop T.C., Victor J.M., Jackson V., van Holde K. The nucleosome family: Dynamic and growing // Structure. 2009. Vol. 17. N 2. P. 160—171. 7. Tomschik M., van Holde K., Zlatanova J. Nucleosome dynamics as studied by single-pair fluorescence resonance energy transfer: A reevaluation // J. Fluoresc. 2009. Vol. 19. N 1. P. 53—62. 8. Hondele M., Ladurner A.G. Catch me if you can how the histone chaperone fact capitalizes on nucleosome breathing // Nucleus-Austin. 2013. Vol. 4. N 6. P. 443—449. 9. Mozziconacci J., Victor J.M. Nucleosome gaping supports a functional structure for the 30 nm chromatin fiber // J. Struct. Biol. 2003. Vol. 143. N 1. P. 72—76. 10. Studitsky V.M., Clark D.J., Felsenfeld G. Overcoming a nucleosomal barrier to transcription // Cell. 1995. Vol. 83. N 1. P. 19—27. 11. Flaus A., Rencurel C., Ferreira H., Wiechens N., Owen-Hughes T. Sin mutations alter inherent nucleosome mobility // EMBO J. 2004. Vol. 23. N 2. P. 343—353. 12. Dror R.O., Dirks R.M., Grossman J.P., Xu H.F., Shaw D.E. Biomolecular simulation: A computational microscope for molecular biology // Annu. Rev. Biophys. 2012. Vol. 41. P. 429—452. 13. Lindorff-Larsen K., Piana S., Palmo K., Maragakis P., Klepeis J.L., Dror R.O., Shaw D.E. Improved side- chain torsion potentials for the amber ff99sb protein force field // Proteins. 2010. Vol. 78. N 8. P. 1950—1958. 14. Guy A.T., Piggot T.J., Khalid S. Single-stranded DNA within nanopores: Conformational dynamics and implications for sequencing; a molecular dynamics simulation study // Biophys. J. 2012. Vol. 103. N 5. P. 1028—1036. 15. Davey C.A., Sargent D.F., Luger K., Maeder A.W., Richmond T.J. Solvent mediated interactions in the structure of the nucleosome core particle at 1.9 C resolution // J. Mol. Biol. 2002. Vol. 319. N 5. P. 1097—1113. 16. Banks D.D., Gloss L.M. Equilibrium folding of the core histones: The h3-h4 tetramer is less stable than the h2a-h2b dimer // Biochemistry. 2003. Vol. 42. N 22. P. 6827—6839. 17. Essmann U., Perera L., Berkowitz M.L., Darden T., Lee H., Pedersen L.G. A smooth particle mesh ewald method // J. Chem. Phys. 1995. Vol. 103. N 19. P. 8577—8593. 18. Bussi G., Donadio D., Parrinello M. Canonical sampling through velocity rescaling // J. Chem. Phys. 2007. Vol. 126. N 1. P. 014101 19. Nose S., Klein M.L. Constant pressure moleculardynamics for molecular-systems // Mol. Phys. 1983. Vol. 50. N 5. P. 1055—1076. 20. Pronk S., Pall S., Schulz R., Larsson P., Bjelkmar P., Apostolov R., Shirts M.R., Smith J.C., Kasson P.M., van der Spoel D., Hess B., Lindahl E. Gromacs 4.5: A high-throughput and highly parallel open source molecular simulation toolkit // Bioinformatics. 2013. Vol. 29. N 7. P. 845—854. 21. Sadovnichy V., Tikhonravov A., Voevodin V., Opanasenko V. “Lomonosov”: Supercomputing at moscow state university // Contemporary high performance computing: from petascale toward exascale / Ed. by S.V. Jeffery. Boca Raton: CRC Press, 2013. P. 283—307. 22. Humphrey W., Dalke A., Schulten K. Vmd: Visual molecular dynamics // J. Mol. Graph. Model. 1996. Vol. 14. N 1. P. 33—38. 23. Shaw D.E. Millisecond-long molecular dynamics simulations of proteins on a special-purpose machine // Biophys. J. 2013. Vol. 104. N 2. P. 45a. Ïîñòóïèëà â ðåäàêöèþ 00.00.2014 CONFORMATIONAL FLEXIBILITY OF NUCLEOSOMES: A MOLECULAR DYNAMICS STUDY G.A. Armeev, K.V. Shaitan, A.K. Shaytan One of the major problems of molecular biology is studying the underlying principles of chromatin work. This problem is associated with understanding the processes of epigenetic regulation of gene expression, DNA repair and heredity mechanisms. The basic unit of chromatin organization is nucleosome, which consists of DNA and histone proteins. For a long time nucleosome structure obtained by X-ray crystallography was considered as the ultimate and main conformational state of nucleosomes, but recent experimental data indicates that chromatin functions depends on conformation state of nucleosomes. Nevertheless, there is no detailed understanding of the conformational dynamics 54 ÂÅÑÒÍ. ÌÎÑÊ. ÓÍ-ÒÀ. ÑÅÐ. 16. ÁÈÎËÎÃÈß. 2015. ¹ 3 of nucleosomes at the atomistic level. In this paper, we have addressed this problem using molecular dynamics simulations of nucleosome in explicit solvent. We have investigated the dynamics of nucleosome on the 500 ns time interval, produced covariance analysis of the trajectory and derived the collective motions, studied the distribution of water molecules in the system and assumed the role of internal water. Key words: nucleosomes, chromatin, molecular dynamics, epigenetics. Ñâåäåíèÿ îá àâòîðàõ Àðìååâ Ãðèãîðèé Àëåêñååâè÷ — àñïèðàíò êàôåäðû áèîèíæåíåðèè áèîëîãè÷åñêîãî ôàêóëüòåòà ÌÃÓ. Òåë.: 8-906-759-56-35; e-mail: armeev@molsim.org Øàéòàí Êîíñòàíòèí Âîëüäåìàðîâè÷ — äîêò. ôèç-ìàò. íàóê, ïðîô. êàôåäðû áèîèíæåíåðèè áèîëîãè÷åñêîãî ôàêóëüòåòà ÌÃÓ. Òåë.: 8-495-939-23-74; e-mail: shaytan49@yandex.ru Øàéòàí Àëåêñåé Êîíñòàíòèíîâè÷ — êàíä. ôèç-ìàò. íàóê, ñò. íàó÷. ñîòð. êàôåäðû áèîèíæåíåðèè áèîëîãè÷åñêîãî ôàêóëüòåòà ÌÃÓ. Òåë.: 8-495-939-57-38; e-mail: alex@molsim.org