Реконструкция регуляции метаболизма метионина
реклама

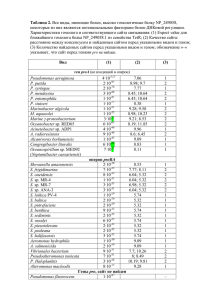
Татьяна Холина, Москва, МГУ ФББ Софья Шерстнева, Москва, школа №463 Инна Суворова, ИППИ РАН Семен Лейн, ИППИ РАН Реконструкция регуляции метаболизма метионина Методы Введение Регуляция метаболизма метионина у бактерий осуществляется факторами транскрипции MetR (LysR семейство) и MetJ. Целью нашей работы было реконструировать регулоны MetR и MetJ в различных таксономических группах, сравнить их между собой и ранее описанными регулонами Enterobacteriales и Shewanellaceae, проследить эволюционные изменения. Результаты Поиск гомологов осуществлялся с помощью программы BLAST. Реконструкция регулонов, поиск ортологов, построение профилей (PWM) осуществлялись при помощи RegPredict. Для каждой таксономической группы был построен собственный профиль. Поиск потенциальных мотивов связывания осуществлялся методом филогенетического футпринтинга. Принадлежность гена к регулону проверялась методом проверки соответствия. Для выравнивания белковых и нуклеотидных последовательностей использовалась программа MUSCLE. Для построения филогенетических деревьев мы пользовались пакетом программ Phylip, а также программой Dendroscope для их визуализации. Диаграммы LOGO были получены при помощи WebLogo. Кроме того, мы пользовались такими базами данных, как NCBI, RegPrecise, KEGG, MicrobesOnline, GLAMM, SEED. Таблица 1. Строение регулонов метаболизма метионина MetJ и MetR MetR Oceanospirillales/Alteromonadales Alteromonadales 2-keto-4-methylthiobutyrate mtnC 2, 3-diketo-5-methylthiopentyl-1-phosphate mtnB mtnD Rhodobacterales 1, 2-dihydroxy-3-keto-5-methylthiopentene S-adenosylmethioninamine MetR and MetJ 5-methylthioribulose-1-phosphate mtnA are two transcription mtnK mtnN 5-methylthioribose-1-phosphate MTR MTA regulators of the methionine metabolism Рис. 1. Схема метаболизма метионина Синим цветом показана регуляция MetJ, красным – MetR, фиолетовым – in bacteria. We have двойная регуляция reconstructed methionine Fig. 1. Scheme of the methionine metabolism regulons in various taxonomic , Blue color denotes MetJ regulation, red – MetR, violet – dual regulation groups. It has been shown that MetJ controls expression of most methionine metabolic genes, but is present in only few taxa, while MetR is found in many different Proteobacteria, but regulates only a few genes. Presumably, those bacteria that lack MetJ have yet unidentified transcription regulators of the methionine metabolism. Moreover, in some Betaproteobacteria and Pseudomonadales we have discovered new LysR-family regulators, homologous to MetR, that control metC and some other methionine metabolic genes. Shewanellaceae Group 2 Psychromonadaceae/ Aeromonadales Group 1 Enterobacteriales Pseudomonadaceae S H E W A N E L L A C E A E Vibrionales Oceanospirillales/Alteromonadales Ralstonia Pasteurellales Burkholderia Рис. 2. Филограммы транскрипционных факторов MetJ и MetR Показаны таксономические группы и диаграммы LOGO мотивов связывания Fig. 2. Phylogenetic trees of MetJ and MetR transcription factors Taxonomic groups and LOGO diagrams of binding motifs are shown A L T E R O M O N A D. + + + + + + + + + + + 0 0 + + + 0 + 0 + + + Salmonella typhimurium + + + 0 + + + + + + + 0 0 + 0 + 0 + 0 + + + Citrobacter koseri + + + 0 + + + + + + + + + + + + + + + + + + Klebsiella pneumoniae + + + + + + + + + + + + + + + + + + + + + + Enterobacter sp. 638 + + + + + + + + + + + + + + + + + + + + + + Erwinia amylovora + + + 0 0 + + + + + + + 0 0 0 + + + 0 + + + Yersinia pestis KIM + + + 0 + + + 0 + + + 0 + + 0 0 0 + 0 + + + Serratia proteamaculans 568 + + + + + + + + + + + + + + + + + + + + + + Erwinia carotovora + + + + + + + + + + + + 0 + + + + + 0 + + + Edwardsiella tarda + 0 + 0 + + + + + + + 0 0 + 0 0 0 + + + + + Proteus mirabilis + + + 0 + + + + + + + 0 0 + 0 0 0 + 0 + + + Photorhabdus luminescens + + + 0 + + + + + + + 0 0 0 0 0 0 + + + + + Haemophilus influenzae + + + + + + + 0 0 + + Aggregatibacter aphrophilus + + + + + + + 0 0 + + Pasteurella multocida + + + + + + + 0 0 + + Mannheimia succiniciproducens + + + + + + 0 + 0 + + Actinobacillus succinogenes + + + + + 0 0 + + + + Haemophilus somnus + + + + + + + 0 0 + + Actinobacillus pleuropneumoniae + + + + + + + + + + + Haemophilus ducreyi + 0 + 0 0 0 0 0 0 + 0 Haemophilus parasuis + + + + + + + 0 0 + + Vibrio cholerae + + + 0 + 0 + + + + + 0 + 0 + + Vibrio vulnificus + + + + 0 + + + + + + + + 0 + + Vibrio harveyi + + + + + + + + + + + + + + + + Vibrio parahaemolyticus + + + + + + + + + + + + + 0 + + Vibrio shilonii + + + + + 0 + + + + + 0 + + + + Vibrio splendidus + + + + + + + + + 0 + + + + + + Vibrio fischeri + + + 0 0 + + + + + + 0 + 0 + + Vibrio salmonicida + + + 0 0 + + + + + 0 0 + 0 + + Vibrio angustum + + + 0 + + + + + 0 0 + + + + + Photobacterium profundum + + + + + + + + + + + 0 + 0 + + Psychromonas ingrahamii 37 + + + 0 0 0 0 + 0 + 0 + + 0 + + Psychromonas sp. CNPT3 + 0 + 0 0 0 0 0 0 0 0 0 0 0 + + Moritella sp. PE36 + + + + + 0 + + 0 0 + + + 0 + + Aeromonas hydrophila + + + + + + + + + + + + + + + + Aeromonas salmonicida + + + + + + + + + + 0 + + + + + Tolumonas auensis + + + 0 0 0 + + + + + + + 0 + + Shewanella oneidensis + + 0 + + + + + + + + + + 0 + + + Shewanella putrefaciens + + + + + + + + + + + + + + + + + Shewanella sp W3-18-1 + + + + + + + + + + + + + + + + + Shewanella sp ANA-3 + + 0 + + + + + + + + + + 0 + + + Shewanella sp MR-4 + + 0 + + + + + + + + + + 0 + + + Shewanella sp MR-7 + + 0 + + + + + + + + + + 0 + + + Shewanella baltica + + + + + + + + + + + + + + + + + Shewanella denitrificans + + 0 + + + + + + + + + + 0 + + + Shewanella frigidimarina + + 0 + + + + + + + + 0 + 0 + + + Shewanella amazonensis + + 0 + + + + + 0 + + + + 0 + + + Shewanella loihica + + 0 + + + + + + + + + + 0 + + + Shewanella pealeana + + 0 + + + + + 0 + + + + 0 + + + Shewanella halifaxensis + + 0 + + + + + + + + 0 + 0 + + + Shewanella piezotolerans + + 0 + + + + + + + 0 0 + 0 + + + Shewanella sediminis + + 0 + + + + + + + + + + 0 + + + Shewanella woodyi + + 0 + + + + + + + + 0 + 0 + + + Pseudoalteromonas atlantica + 0 0 + 0 + 0 0 + 0 + 0 0 0 0 + + + Alteromonas macleodii + 0 0 0 0 + 0 0 0 0 + + + + 0 + + + Glaciecola sp. HTCC2999 + 0 0 0 0 + 0 0 + 0 + + + + 0 + + + Colwellia psychrerythraea + + +* + + + 0 + + + 0 0 + 0 0 + + + Alteromonadales bacterium + + + + + + 0 + + + 0 + + + 0 + + + Pseudoalteromonas haloplanktis + + + + + + 0 + + + 0 + + + + + + + Pseudoalteromonas tunicata + + + + + + 0 + 0 + 0 + + + + + + + Idiomarina baltica + 0 0 + + 0 + 0 + 0 0 0 0 0 0 + + 0 Idiomarina loihiensis + 0 +* + + 0 + 0 + 0 0 0 0 0 0 + + 0 + thrABC mtnE Burkholderia P S Y C H R. asd metT S-adenosylmethionine folE folE GTP Methionine metK luxS metNIQ glyA TH F mccBA mmuM glyA Serine S-methylmethionine 5-methyl-THF metK 5,10-methylene-THF mtnABCDK Glycine Vibrionales mtnE Shewanellaceae metF/metF2 mmuM metE/metE2 metH mmuP Cobalt metE2-2 Vitamin B12 metE2 metH btuB mtsABC metE S-adenosylhomocysteine Homocysteine metF Cystathionine metF-II luxS+mtnN metC metC metB mccBA V I B R I O N A L E S metY metY O-acetylhomoserine metB O-succinyhomoserine P A S T E U R E L L A L E S metX Pasteurellales metA Threonine metX metA Cystein Alteromonadales metL Homoserine thrBC E N T E R O B A C T E R I A L E S mtsABC metL, thrA Escherichia coli Genomes/Regulated genes metT Aspartate-4-semialdehyde btuB Psychromonadaceae/Aeromonadales mmuP 4-phospho-aspartate asd Enterobacteriales metNIQ MetJ metL, thrA metR Aspartate metJ Мы реконструировали регулоны MetR и MetJ для всех выбранных групп бактерий и выявили их мотивы связывания. Было показано, что MetR встречается во многих семействах Proteobacteria, но узкоспецифичен и регулирует малый участок метаболической сети. В то же время MetJ был обнаружен только среди Gammaproteobacteria, однако регулирует большую часть генов метаболизма метионина. При этом в данных таксонах MetR, как правило, находится под регуляцией MetJ. Отсутствие MetJ во многих группах бактерий позволяет предположить наличие других регуляторов метаболизма метионина. Кроме того, в ряде Betaproteobacteria и Pseudomonadaceae были обнаружены два близких LysR-регулятора, гомологичных MetR. Для них также были идентифицированы мотивы связывания и реконструированы регулоны. В состав обоих регулонов входили гены metC, а также еще одной цистатион бета-лиазы. У представителей Pseudomonadaceae регулон также включал гены аспартат аминотрансферазы и ABC-транспортера аминокислот. Синим цветом показана регуляция MetJ, красным – MetR, фиолетовым – двойная регуляция; + – ген присутствует в геноме, 0 – отсутствие ортолога Table 1. Structure of the MetJ and MetR regulons of methionine metabolism Blue color denotes MetJ regulation, red – MetR, violet – dual regulation; + the gene is present, 0 – no orthologous genes in genome