Лекция 12 - DNA punctuation
реклама

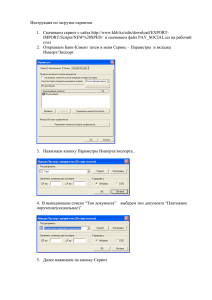
Практическое занятие Нахождение гомологов, выравнивание последовательностей, построение и статистическая оценка филогенетического дерева 1. Отобрать 15-20 гомологичных последовательностей • По выбору, например: – – – – – – – – 16S RNA 50S RNA tRNAs (для каждой аминокислоты своя tRNA) ATP synthase (alpha, beta, gamma, delta, epsilon subintis) tRNA synthases ((для каждой аминокислоты своя) DNA polymerase (alpha and beta subunits) RNA polymerase Ribosomal proteins (их ~50, так что следите за номером, буква L – large или S - small субъединицы рибосомы) – Любой ген, который вы хотите проанализировать 1. Отобрать 15-20 гомологичных последовательностей • • Идете на сайт http://www.ncbi.nlm.nih.gov/ Среди баз данных выбираете либо protein, либо nucleotide. Например: Измените формат на FASTA Скопируйте в текстовый файл Идем в BLAST В зависимости от того, какие вы гены выбрали, кодирующие или РНК Вставпоследова Удобно, чтобы результаты высвечивались в новом окне Или даже 5000,10000 Можно выбрать любую матрицу, запомните какую. Прокрутите результаты вниз, до начала выравниваний. Начинайте отбирать последовательности снизу вверх, чтобы они были <=80% идентичности Щелкните на последовательность, это вас перенесет на локальное выравнивание. Соберите 15-20 последовательностей с условием, что их % identity < 80 Сохраните все по в один текстовый файл 2. Выровнять отобранные последовательности Скачайте программу множественного выравнивания Скачиваем Clustalx (графический вариант) – есть дистрибутивы для всех систем Ваше выравнивание не должно быть таким хорошим, так как вы должны были выбрать более разошедшиеся последовательности. 2. Построить филогенетическое дерево методом ближайшего соседа (NJ) и выполнить bootstrap-анализ Все в той же программе ClustalX Формат phylip читается почти всеми программами отображения деревьев (TreeViewers) Change Label To Node here Скачиваем какой-нибудь Tree Viewer TreeView под Windows Открываете phb (bootstrapped) файл FigTree или TreeViewer или в любой другой программе визуализации деревьев В FigTree – появится сообщение Нажмите Ok Чтобы увидеть бутстрэп значения, надо в Branch Labels в разделе Display выбрать label Bootstrap-значения должны быть в диапазоне от 1 до 1000 (мы так задали). Могли бы задать от 1 до 100. Отчетность Распечатать или послать по email: 1. Текстовый файл с отобранными последовательностями. Указать использованные параметры BLAST (какая матрица была использована, сколько последовательностей было выбрано для поиска) 2. Скриншот выравнивания 3. Скриншот дерева, на котором видны бутстрэп-значения