Cравнение аминокислотных последовательностей
реклама
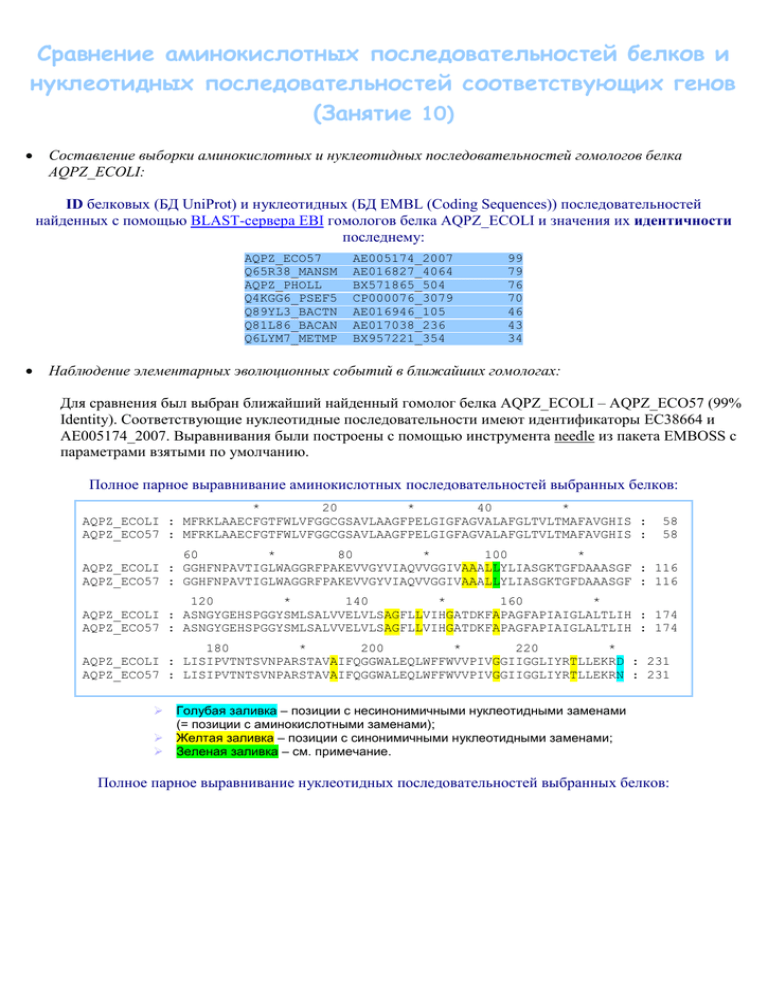
Cравнение аминокислотных последовательностей белков и нуклеотидных последовательностей соответствующих генов (Занятие 10) Составление выборки аминокислотных и нуклеотидных последовательностей гомологов белка AQPZ_ECOLI: ID белковых (БД UniProt) и нуклеотидных (БД EMBL (Coding Sequences)) последовательностей найденных с помощью BLAST-сервера EBI гомологов белка AQPZ_ECOLI и значения их идентичности последнему: AQPZ_ECO57 Q65R38_MANSM AQPZ_PHOLL Q4KGG6_PSEF5 Q89YL3_BACTN Q81L86_BACAN Q6LYM7_METMP AE005174_2007 AE016827_4064 BX571865_504 CP000076_3079 AE016946_105 AE017038_236 BX957221_354 99 79 76 70 46 43 34 Наблюдение элементарных эволюционных событий в ближайших гомологах: Для сравнения был выбран ближайший найденный гомолог белка AQPZ_ECOLI – AQPZ_ECO57 (99% Identity). Соответствующие нуклеотидные последовательности имеют идентификаторы EC38664 и AE005174_2007. Выравнивания были построены с помощью инструмента needle из пакета EMBOSS с параметрами взятыми по умолчанию. Полное парное выравнивание аминокислотных последовательностей выбранных белков: * 20 * 40 * AQPZ_ECOLI : MFRKLAAECFGTFWLVFGGCGSAVLAAGFPELGIGFAGVALAFGLTVLTMAFAVGHIS : AQPZ_ECO57 : MFRKLAAECFGTFWLVFGGCGSAVLAAGFPELGIGFAGVALAFGLTVLTMAFAVGHIS : 58 58 60 * 80 * 100 * AQPZ_ECOLI : GGHFNPAVTIGLWAGGRFPAKEVVGYVIAQVVGGIVAAALLYLIASGKTGFDAAASGF : 116 AQPZ_ECO57 : GGHFNPAVTIGLWAGGRFPAKEVVGYVIAQVVGGIVAAALLYLIASGKTGFDAAASGF : 116 120 * 140 * 160 * AQPZ_ECOLI : ASNGYGEHSPGGYSMLSALVVELVLSAGFLLVIHGATDKFAPAGFAPIAIGLALTLIH : 174 AQPZ_ECO57 : ASNGYGEHSPGGYSMLSALVVELVLSAGFLLVIHGATDKFAPAGFAPIAIGLALTLIH : 174 180 * 200 * 220 * AQPZ_ECOLI : LISIPVTNTSVNPARSTAVAIFQGGWALEQLWFFWVVPIVGGIIGGLIYRTLLEKRD : 231 AQPZ_ECO57 : LISIPVTNTSVNPARSTAVAIFQGGWALEQLWFFWVVPIVGGIIGGLIYRTLLEKRN : 231 Голубая заливка – позиции с несинонимичными нуклеотидными заменами (= позиции с аминокислотными заменами); Желтая заливка – позиции с синонимичными нуклеотидными заменами; Зеленая заливка – см. примечание. Полное парное выравнивание нуклеотидных последовательностей выбранных белков: * 20 * 40 * EC38664 : atgttcagaaaattagcagctgaatgttttggtactttctggcttgtttttggtggct : AE005174_2 : atgttcagaaaattagcagctgaatgttttggtactttctggcttgtttttggtggct : 58 58 60 * 80 * 100 * EC38664 : gtggtagtgctgtactggccgcaggcttcccggaattaggcattggttttgccggcgt : 116 AE005174_2 : gtggtagtgctgtactggccgcaggcttcccggaattaggcattggttttgccggcgt : 116 120 * 140 * 160 * EC38664 : ggcgttggcgttcggtctgaccgttctgacgatggcctttgctgttggtcatatttct : 174 AE005174_2 : ggcgttggcgttcggtctgaccgttctgacgatggcctttgctgttggtcatatttct : 174 180 * 200 * 220 * EC38664 : ggtggtcattttaacccggcggtcactattggtttatgggctggcggacgttttccgg : 232 AE005174_2 : ggtggtcattttaacccggcggtcactattggtttatgggctggcggacgttttccgg : 232 240 * 260 * 280 * EC38664 : caaaagaagtcgttggctacgtaattgcccaggttgtcggcggtattgttgcagcggc : 290 AE005174_2 : caaaagaagtcgttggctacgtaattgcccaggttgtcggcggtattgttgcggcagc : 290 300 * 320 * 340 EC38664 : gctcgtgtatttaattgccagtggtaaaacgggttttgacgcggcagccagcggtttt : 348 AE005174_2 : gctgctgtatttaattgccagtggtaaaacgggttttgacgcggcagccagcggtttt : 348 * 360 * 380 * 400 EC38664 : gcttctaacggttatggcgagcattcaccaggcggttattccatgctttccgcgctgg : 406 AE005174_2 : gcttctaacggttatggcgagcattcaccaggcggttattccatgctttccgcgctgg : 406 * 420 * 440 * 460 EC38664 : tagttgaactggtattgagtgcaggtttcctgttggtgatccacggcgcaaccgacaa : 464 AE005174_2 : tagttgaactggtattgagtgcgggcttcctgttagtgatccacggtgcaaccgacaa : 464 * 480 * 500 * 520 EC38664 : attcgcgccggcaggttttgcgccgatcgctattggtctggccttaaccctgattcac : 522 AE005174_2 : attcgctccggcaggttttgcgccgatcgctattggtctggccttaaccctgattcac : 522 * 540 * 560 * 580 EC38664 : ttaattagtattccggtgactaacacttctgttaacccggcgcgcagcaccgcggttg : 580 AE005174_2 : ttaatcagtattccggtgactaacacttctgttaacccggcgcgcagcaccgcggttg : 580 * 600 * 620 * 6 EC38664 : ctatcttccagggcggctgggcattagaacaactgtggttcttctgggtggtgccaat : 638 AE005174_2 : ccatcttccagggcggctgggcattagaacaactgtggttcttctgggtggtgccaat : 638 40 * 660 * 680 * EC38664 : tgtcggcggcattatcggtggtctgatttaccggaccctgctggaaaagcgtgattaa : 696 AE005174_2 : tgtcggtggcattatcggtggtctgatttaccggactctgctggaaaagcgtaattaa : 696 Голубая заливка – кодоны с несинонимичными нуклеотидными заменами; Желтая заливка – кодоны с синонимичными нуклеотидными заменами; Зеленая заливка – см. примечание; Красный шрифт – непосредственно позиция с заменой. Примечание: позиция 295 в нуклеотидном выравнивании имеет ряд особенностей. Так, при расшифровке кодонов с использованием таблицы стандартного генетического кода выясняется, что в аминокислотном выравнивании на соответствующем месте (позиция 99) должны стоять валин для белка AQPZ_ECOLI и лейцин – для AQPZ_ECO57, в то время как на самом деле там стоят лейцины в обоих случаях. Поскольку выравнивания не содержат гэпов (и следовательно номера позиций в них совпадают с номерами позиций в самих последовательностях), а генетический код E.coli не отличается от стандартного можно сделать вывод, что эти различия являются результатами ошибки в базе данных. Так как Swiss-Prot в отличие от EMBL(CDS) является курируемой БД, позиции 295/99 я посчитал содержащими синонимичную нуклеотидную замену. Сравнение двух полных парных выравниваний: аминокислотных последовательностей: Белки (ID): AQPZ_ECOLI AQPZ_ECO57 Identity: Число замен: 99% 1 из 231 Замены: Позиция: Значение: D (Asp) 231 N (Asn) нуклеотидных последовательностей: Гены (AC): Identity: Число замен: Позиция: Значение: A G 286 Позиция: 295 Позиция: 429 Позиция: EC38664 97% AE005174_2007 14 из 696 441 Позиция: 471 Позиция: 582 Позиция: 675 Замены: Кодон: Позиция: 3 289 Значение: C G Кодон: Позиция: 3 296 Значение: A G Кодон: Позиция: 3 432 Значение: G A Кодон: Позиция: 3 453 Значение: G T Кодон: Позиция: 3 528 Значение: T C Значение: C T Кодон: Позиция: 3 645 Кодон: Позиция: 3 691 Значение: G A Кодон: Значение: G C Кодон: Значение: T C Кодон: Значение: C T Кодон: Значение: T C Кодон: Значение: C T Значение: G A Кодон: 3 1 3 3 3 3 Кодон: 1 В пункте “Кодон” указана позиция замены в кодоне; Красным шрифтом выделены позиции с заменой в третьем нуклеотиде кодона; Синим шрифтом выделены позиции с транзитивной заменой. Матрица замен нуклеотидного выравнивания: A A T G C 0 5 0 T 1 6 G 2 Как можно заметить в большинстве случаев (6 к 1) нуклеотидные замены происходили в третьей позиции кодона. Это можно обьяснить особенностями вырожденности генетического кода: для большинства аминокислот она наблюдается именно в третьей позиции, что создает наибольшую вероятность заменам в этой позиции стать синонимичными и, следовательно, не быть элиминированными в процессе эволюции стабилизирующим отбором. Так же велика (11 к 3) доля транзитивных мутаций, т.е. замен пурина на пурин (A=>G, G=>A) или пиримидина на пиримидин (T=>C, C=>T), по сравнению с трансверсионными, эта зависимость, как известно, носит общий для всего живого характер и связана со структурной схожестью пуринов и пиримидинов между собой. Соотношение числа несинонимичных и синонимичных замен нуклеотидов равно 1/13 ≈ 0,08. Известно, что чем меньше это отношение, тем выше было давление стабилизирующего отбора в процессе эволюции исследуемых белков. Можно предположить, что высокая степень консервативности аминокислотной последовательности (следствие повышенного давления стаб. отбора), свойственная моему белку, связана с выполняемой им функцией в качестве трансмембранного транспортера и существующей необходимостью поддержания структурной консервативности. Исследование зависимости процента совпадений последовательностей генов от процента совпадений последовательностей соответствующих белков: Текст скрипта для определения попарной идентичности последовательностей: gsn1=0 gsn2=0 echo "1st column - gene identity, 2nd column - protein" > scriptresult while [ $gsn1 -le "7" ] do let gsn2=$gsn1+1 while [ $gsn2 -le "7" ] do geneident=`needle GS"$gsn1" GS"$gsn2" -gapopen 10 -gapextend 0.5 -auto stdout | grep "# Identity:" | fold -w 25| grep "%" | fold -w 4 | grep -v "%" | tr "." ","` protident=`needle PS"$gsn1" PS"$gsn2" -gapopen 10 -gapextend 0.5 -auto stdout | grep "# Identity:" | fold -w 25| grep "%" | fold -w 4 | grep -v "%" | tr "." ","` echo "$gsn1" and "$gsn2" $geneident $protident >> scriptresult let gsn2=$gsn2+1 done let gsn1=$gsn1+1 done Имена файлам последовательностей были даны по их номерам в порядке уменьшения идентичности белку AQPZ_ECOLI (последнему был присвоен номер “0”) c добавлением указателя GS или PS – Protein/Gene Sequence. На момент написания данного скрипта я имел довольно смутное представление о использовании регулярных выражений, поэтому вытаскивание из стандартной выдачи needle непосредственно значений Identity пришлось производить подручными средствами – последовательным разбиением строки на две, используя команду fold, и отбором необходимой строки из получившихся командой grep. Результаты выполнения скрипта: 1st column - gene alignment 0 and 1 0 and 2 0 and 3 0 and 4 0 and 5 0 and 6 0 and 7 1 and 2 1 and 3 1 and 4 1 and 5 1 and 6 1 and 7 2 and 3 2 and 4 2 and 5 2 and 6 2 and 7 3 and 4 3 and 5 3 and 6 3 and 7 4 and 5 4 and 6 4 and 7 5 and 6 5 and 7 6 and 7 identity, 2nd column - protein 98,0 99,6 70,3 78,4 69,8 76,2 64,5 68,8 50,7 45,2 51,1 43,6 47,8 33,2 72,1 77,9 69,9 76,2 62,5 68,2 49,5 45,2 51,4 43,6 49,1 33,5 67,4 73,2 61,4 66,8 54,6 44,6 53,5 44,0 52,5 32,8 57,5 60,9 52,0 45,6 53,3 41,6 50,1 35,6 51,2 43,9 45,6 42,5 42,2 34,1 52,6 49,4 49,3 32,9 49,1 34,7 По данным, полученным выше, был построен график зависимости Identity генов от Identity соответствующих им белков. Ниже приводится сравнение данного графика с графиком, предложенным в задании и отображающим связь процентов совпадений последовательностей для белка-предшественника гемагглютинина у разных штаммов вируса гриппа. Особые точки, отмеченные на нижеприведенных графиках, представляют два случая: (100,100) – полной (как генной, так и аминокислотной) идентичности, и (5,25) – математическое ожидание (среднее значение) идентичностей двух случайных последовательностей. Таким образом, прямая, их соединяющая представляет собой некую “диагональ” или среднюю линию – усредненную зависимость идентичностей последовательностей (при этом хочется отметить, что с точки зрения биологии, эта линия имеет хоть какой-либо смысл только когда по оси X находится идентичность генов, а по оси Y – белков, так как из полного совпадения нуклеотидной последовательности следует полное совпадение аминокислотной, но не наоборот!). Графики связи процента совпадений последовательностей генов с процентом совпадений последовательностей соответствующих им белков: для гомологов белка AQPZ_ECOLI: 100 для белков-предшественников гемагглютинина разных штаммов вируса гриппа: 100 G e n e 90 80 90 80 I d e n t i t y 70 60 50 40 G e n e I d e n t i t y 70 60 50 40 30 30 20 20 10 10 Protein Identity: Protein Identity: 0 0 0 10 20 30 40 50 60 70 80 90 100 0 10 20 30 40 50 60 70 80 Темно-синие линии – графики зависимостей; Красные линии – линии тренда (экспоненциальная зависимость); Розовые линии – прямые, соединяющие точки (5,25) и (100,100) (“средние” линии). Сравнение вышеприведенных графиков (отображены только линии тренда): 100 90 80 70 60 50 40 30 20 G e n e I d e n t i t y 90 100 Синяя линия – тренд графика зависимости для гомологов белка AQPZ_ECOLI; Оранжевая линия – тренд графика зависимости для белков-предшественников гемагглютинина; Розовая линия – прямая, соединяющая точки (5,25) и (100,100) (“средняя” линия). Как можно заметить оба графика имеют как сходства, так и различия. Общим для них является то, что аппроксимирующая кривая (линия тренда) на отрезке от 30 до 100 лежит ниже средней линии, а при ее продолжении к X=5 начиная с X≈30 лежит выше. Конечно данное наблюдение довольно условно, и реальная зависимость идентичностей в области от 0 до 30 может вести себя как угодно, но данный предполагаемый факт имеет довольно логичное объяснение. При высокой схожести белковых последовательностей, нуклеотидные имеют меньшую идентичность за счет скрытых синонимичных мутаций, но при уменьшении Identity белков, на фоне увеличения числа несинонимичных мутаций значение этих “резервов” снижается, и доминирующим становится влияние фактора длины – чем длиннее последовательность, тем менее равное число мутаций влияет на идентичность (что вполне логично следует из формулы Identity=[число совпадений]/[общая длина выравнивания]), а так как любая нуклеотидная последовательность в три раза длиннее соответствующей аминокислотной, схожесть генов становится выше схожести белков. Различия графиков хорошо видны на общей таблице – аппроксимация (будем учитывать лишь интервал от X=100 до пересечения двух трендов, т.к. как было сказано выше, в дальнейшем сложно с уверенностью предсказать поведение зависимости) графика для гомологов белка AQPZ_ECOLI лежит ниже чем линия тренда предшественников агглютинина. Из этого наблюдения можно сделать вывод о большей консервативности на белков моей выборки, так как меньшее сходство генов по сравнению с белками говорит о большом числе накопленных синонимических мутаций, что является косвенным свидетельством повышенного давления стабилизирующего отбора. Большая консервативность гомологов белка AQPZ_ECOLI вполне предсказуема, ведь, как известно, скорость мутирования вирусных геномов наиболее высока среди всех форм жизни.