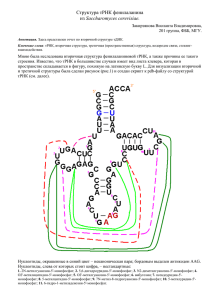
Отчет о вторичной структуре треониновой тРНК
реклама

Название: Структура Треонин-тРНК Esherichia coli. Автор: Шпильман А.А. 202 ФББ МГУ Аннотация: изучение вторичной и третичной структуры транспортной РНК (тРНК) различными методами биоинформатики Ключевые слова: тРНК, 1qf6. Введение: Основной задачей тРНК является доставка к рибосоме активных остатков аминокислот и обеспечивать их включение в синтезирующуюся белковую цепь. Вторичная структура любой тРНК напоминает клеверный лист, а в пространстве молекула тРНК похожа на букву L . Целью настоящей работы стало изучение взаимодействий за счет которых образуются вторичная и третичная структуры треонин-тРНК кишечной палочки. Результаты: Схема вторичной структуры: A C C A G–C C–G C-G G-C A-U U-A A-U DhuGACUCGAU UAUCCUCAG Dhu | | | | | | | | | C GGDhuAGAGCA-AG CGUAGG5muPsu G-A G7mU C-G G-C C-G A-U U A U Aet CGU Обеспечивание третичной структуры в основном водородными связями: 8(U)-14(A), 15(G)-48(С) ,56(С)-19(G), (отмечено зеленым, синим и розовым) Схема Вторичной структуры тРНК, полученная при помощи алгоритма Зукера. Обсуждение: 1)Пространственная форма тРНК образуется за счет взаимодействий между T- и Dпетлями, которые сближаются и скрепляются между собой посредством образования дополнительных, часто необычных пар оснований. 2)Отличия в прдесказаниях состоят в том, что mfold не провел связь между нестандартными с не каноническим взаимодеиствием, поскольку алгоритм Зукера стремится свести к минимуму кол-во неканонических пар. 3)тРНК выполняет две основных функции: акцепторную – способность ковалентно связываться с аминоацильным остатком, превращаясь в аминоацил-тРНК, и адапторную – способность узнавать триплет генетического кода, соответствующий транспортируемой аминокислоте, и обеспечивать поступление аминокислоты на законное место в растущей цепи белка. Но реакция должна быть катализирована специальным ферментом аминоацилтРНК-синтетазой, которая узнает «свою» тРНК в основном за счет структурного взаимодействия. Таким образом, структура тРНК крайне важна для ее функционирования. Сопроводительные материалы: В файле 1qf6.spt содержится скрипт для Rasmol, позволяющий визуализировать основные элементы структуры тРНК. Материалы и методы: 3D структура тРНК получена в Protein Data Bank(1). ID: 1QF6 Комплементарность пар определена программой find_pair пакета 3DNA. Результаты обработаны с помощью Word и RasMol. Литература: 1) Protein Data Bank (http://www.rcsb.org/pdb/cgi/explore.cgi?pid=60551096549937&pdbId=1IL2