Ключевые слова.
реклама

Материалы к занятию 27
Ключевые слова.
Филогенетическое дерево (phylogenetic tree). Построенное, реконструированное (предполагаемое, inferred)
дерево.
Термины для описания дерева: узел, вершина (node), лист (OTU), корень(root), ребро или ветвь(edge, branch),
топология, операционные таксономические единицы (operational taxonomic unit, OTU ).
Свойства деревьев: бинарное, разрешенное и неразрешенное, корневое и бескорневое, ультраметрическое,
аддитивное.
Алгоритмы, основанные на матрицах попарных эволюционных расстояний.
UPGMA (Unweighted Pair Group with Arithmetic Mean или нечто вроде «попарной невзвешенной кластеризации
с арифметическим усреднением»). Метод ближайших соседей (Neighbor-joining, NJ).
Иные алгоритмы: максимальная экономия (бережливость, MP=maximal parsimony), максимальное
правдоподобие(ML=maximum likehood)
Задания
Результаты заданий надо красиво оформить в виде файла my_results27.doc. В отчете должно быть
пунктов, названия пунктов – это названия заданий, см. ниже.
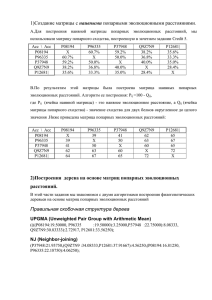
1. Создание матрицы с наивными попарными эволюционными расстояниями.
При выполнении зачетного задания №5 Вы создали матрицу попарного сходства для выборки из 5
гомологичных белков. (см. файл credit5_data.doc в своей директории Credit5).
Преобразуйте эту матрицу в матрицу наивных попарных эволюционных расстояний.
В отчете приведите обе матрицы и опишите выбранное Вами правило преобразования.
Матрицу наивных попарных эволюционных расстояний сохраните в виде текстового файла my_prot.dist
следующего формата:
2. Два алгоритма построения дерева на основе матриц попарных эволюционных расстояний.
Программа eneighbor из пакета программ EMBOSS может строить деревья, используя как алгоритм UPGMA,
так и алгоритм NJ.
Для того, чтобы воспользоваться этой программой, зайдите с помощью программы putty на сервер
kodomo.cmm.msu.ru, используйте протокол SSH (SSH-протокол является защищенной версией telnetпротокола). Для того, чтобы найти путь к этой программе, наберите команду:
export PATH="/home/preps/golovin/progs/emboss/bin:${PATH}"
2.а. Запустите программу eneighbor, на вход подайте файл my_prot.dist. Соглашайтесь со всеми заданными
Вам вопросами, тогда будет использован алгоритм NJ. Выходные файлы outfile и outtree сохраните с
расширением nj.
Внимание! Скорее всего программа сообщит о неверных символах, это связано с различиями
синтаксиса Windows и Unix. Откройте файл my_prot.dist. с помощью mcedit и уберите символы, которые
Вам покажутся лишними.
2.б.Запустите программу eneighbor, на вход подайте файл my_prot.dist. Для того, чтобы программа
использовала алгоритм UPGMA, ответьте "N" на вопрос “Neighbor-joining or UPGMA tree”? Выходные
файлы outfile и outtree сохраните с расширением upg .
В отчете укажите полные названия алгоритмов, для каждого приведите правильную скобочную структуру
дерева (см. файлы outtree.*).
3. Визуализация деревьев и сравнение их топологии
3.а. Запустите программу TreeView и откройте в ней файл outtree upg и рассмотрите дерево в различных
формах (для переключения между различными формами служат 4 кнопочки слева вверху).
3.б. Не закрывая окошко с первым деревом, откройте файл outtree nj. Рассмотрите оба дерева в
форме укорененной кладограммы (четвертая кнопка слева наверху окошка). Сохраните оба
изображения в виде графических метафайлов (*.msf).
3.в. Вставьте (как объект) сохраненные изображения деревьев в Ваш отчет. Добейтесь того, чтобы
они стояли рядом и были разумного размера. Под картинками опишите, как происходила
эволюция согласно дереву outtree upg («от самого древнего общего предка произошли белок Х и
общий предок всех остальных, от последнего…….») . Сравните с тем, что показывает дерево outtree nj.
Результаты сравнения запишите!!
В директории public_html создайте поддиректорию doc. Поместите туда Ваш файл my_results27.doc.
На страничке «Второй семестр» (term2.htm) Вашего сайта добавьте к списку гиперссылку «Деревья»,
указывающую на Ваш файл.
Дополнительные задания (для желающих получить бонусные баллы)
1. Пометьте на изображении бескорневого дерева outtree.nj то место, которое соответствует корню
этого дерева, если его рассматривать в форме укорененной кладограммы. Добавьте как отдельную
картинку в Ваш отчет (1 балл)
2. **Проверьте, какое из 2-х полученных деревьев является ультраметрическим, а какое аддитивным.
(см. файлы outfile.*). Ваше мнение должно быть подкреплено конкретными расчетами.
(1.5 балла)
3. На основе Вашей матрицы попарных расстояний постройте «вручную» дерево по алгоритму
UPGMA (по образцу в презентации). В отчете приведите все этапы построения, а также нарисуйте
дерево, укажите длины всех ребер. Сравните с деревом, построенным программой outtree upg
( 3 балла).
4. Сформулируйте запрос для SRS, который позволил бы сразу получить файл со всеми 5-тью
нужными Вам последовательностями. Сам запрос (Query "(((([…..) скопируйте в файл отчета.
С помощью программы ClustalW (ClustalX) постройте бутстрепное дерево ( Boostrap
Tree) для 5-ти гомологов Вашей выборки. Оцените надежность полученной топологии.
Изображение дерева и обсуждение надежности точек ветвления приведите в своем отчете (3
балла)